 首页>
生物试剂
首页>
生物试剂
pAOX1-MCS-His-Amp&Zeo是碧云天研发的以毕赤酵母(Pichia pastoris)为表达菌株,表达C端带有His标签的目的蛋白的真核表达质粒。在多克隆位点(Multiple cloning sites, MCS)按照读码框插入不带有终止密码子的目的基因,就可以表达C端带有His标签的目的蛋白。
毕赤酵母作为蛋白真核表达系统有很多优势:易于操作,蛋白正确折叠以及翻译后修饰等。与昆虫表达系统和哺乳动物细胞表达系统相比,它快速、高效且经济,通常外源蛋白还具有较高的表达水平。与酿酒酵母(Saccharomyces cerevisiae)相比,其外源蛋白表达水平往往高10-100倍;它们均能进行分泌蛋白的N-连接糖苷键修饰,表达分泌蛋白进行糖基化修饰时糖链长度是有差别的,毕赤酵母修饰的分泌蛋白的糖链长度通常是8-14甘露糖残基(Mannose residue),而酿酒酵母修饰的分泌蛋白的糖链长度通常是50-150个甘露糖残基;毕赤酵母很少对分泌蛋白进行O-连接糖苷键的修饰。
本质粒转化X-33, SMD1168H或GS115菌株后,因为宿主菌有功能完好的组氨酸脱氢酶基因,适合进行Zeocin筛选转化子,并能使目的基因在甲醇诱导后实现高水平表达。常用的毕赤酵母宿主菌GS115 (D0412)和KM71 (D0413),二者的组氨酸脱氢酶基因(his4)发生突变,阻止它们合成组氨酸,因此具有His4-营养缺陷标记,适合在组氨酸缺少的培养基进行转化子筛选。GS115/X-33/ SMD1168H菌株具有功能完整的AOX1基因,属于Mut+ (Methanol utilization plus)表型,即甲醇利用正常,在甲醇诱导的情况下生产快速。KM71/KM71H菌株的AOX1位点被ARG4基因插入破坏,表型为Muts (Methanol utilization slow),即甲醇利用缓慢,在甲醇诱导的情况下生产缓慢。
本质粒5'端含有醇氧化酶基因AOX1启动子,可以高效启动目的基因的毕赤酵母胞内表达。在毕赤酵母中,AOX1启动子属于甲醇诱导型强启动子,其严格依赖于甲醇的诱导,并受葡萄糖、果糖、甘油、乙醇等碳源的抑制,因此可通过控制葡萄糖、甘油等碳源的消耗和甲醇的流加来调控AOX1启动子的表达。
本质粒为氨苄青霉素(Ampicillin)和博来霉素(Zeocin)双抗性。大肠杆菌博来霉素筛选推荐浓度为25-50μg/ml (低盐LB培养基,NaCl浓度不能超过5g/L),酵母筛选推荐浓度为50-300μg/ml (YPD或基本培养基)。氨苄青霉素通常配制成100mg/ml的储备液,使用时可以按照1:1000的比例稀释使用,即推荐使用浓度为100μg/ml。
pAOX1-MCS-His-Amp&Zeo质粒(4231bp)的图谱如下:
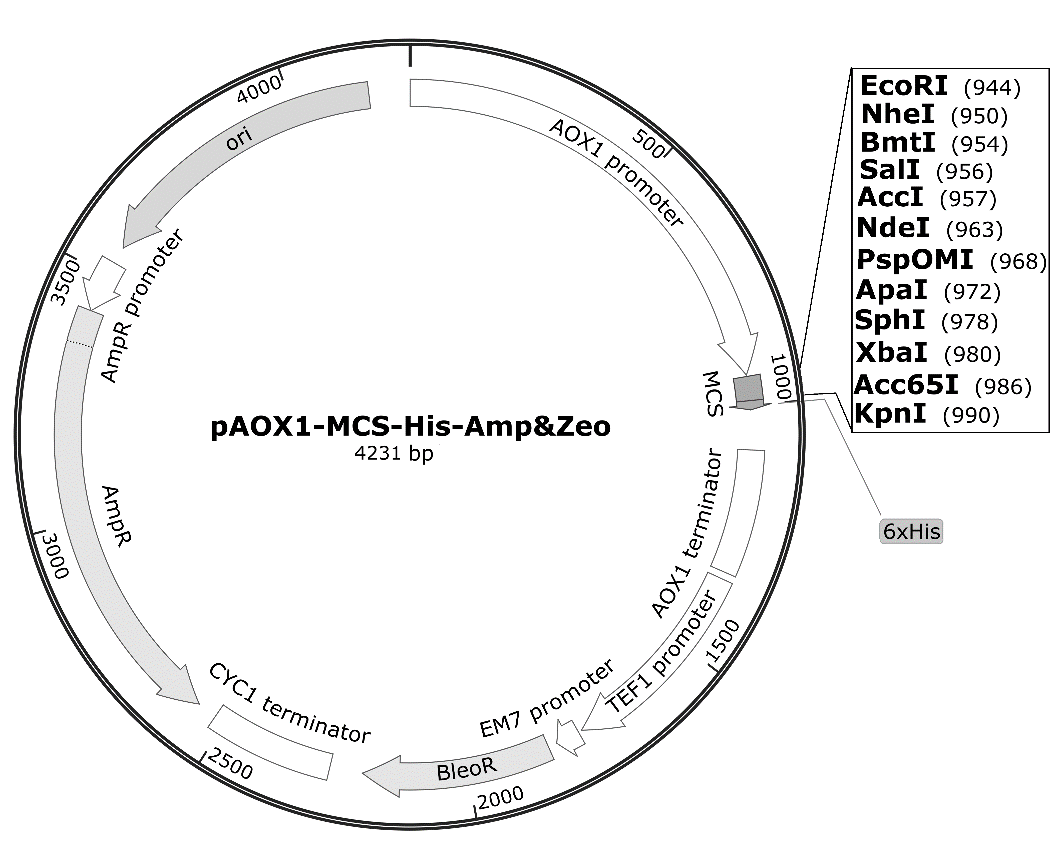
pAOX1-MCS-His-Amp&Zeo质粒的主要信息如下:
| Feature Nucleotide | Position |
| AOX1 promoter | 2..940 |
| 6×His | 992..1009 |
| AOX1 terminator | 1089..1335 |
| TEF1 promoter | 1350..1761 |
| EM7 promoter | 1769..1816 |
| BleoR | 1835..2209 |
| CYC1 terminator | 2275..2522 |
| AmpR | 2563..3423 |
| AmpR promoter | 3424..3528 |
| ori | 3563..4151 |
pAOX1-MCS-His-Amp&Zeo的详细图谱见说明书:https://www.beyotime.com/Manual/D2883 pAOX1-MCS-His-Amp&Zeo.pdf
pAOX1-MCS-His-Amp&Zeo中没有的酶切位点包括:
| AanI | AarI | AbsI | AccIII | Acc36I | AcvI | AfeI |
| AflII | AhlI | AjuI | AloI | Aor13HI | Aor51HI | ArsI |
| AscI | AsiSI | AspI | AspA2I | AvrII | AxyI | BaeI |
| BanIII | BarI | BbeI | BbrPI | BbvCI | BcuI | BfrI |
| BfuAI | BlnI | BoxI | Bpu10I | Bsa29I | BsaAI | Bse21I |
| BseAI | BseCI | BsgI | BshVI | BsmBI | Bsp13I | Bsp68I |
| BspDI | BspEI | BspMI | BspQI | BspTI | BspXI | BssNAI |
| Bst98I | Bst1107I | BstAFI | BstAPI | BstBAI | BstENI | BstHPI |
| BstPAI | BstSNI | BstZ17I | Bsu15I | Bsu36I | BsuTUI | BtuMI |
| BveI | CciNI | Cfr42I | ClaI | CpoI | CspI | CspCI |
| DinI | Eco47III | Eco72I | Eco81I | Eco105I | EcoNI | EgeI |
| EheI | Esp3I | FalI | FspAI | HpaI | I-CeuI | I-PpoI |
| I-SceI | KasI | Kpn2I | KspI | KspAI | LguI | Mly113I |
| MreI | MroI | MspCI | NarI | Nb.BbvCI | Nb.Bpu10I | NotI |
| NruI | Nt.BbvCI | Nt.Bpu10I | Nt.BspQI | PacI | PaeR7I | PalAI |
| PaqCI | PasI | PciSI | PflFI | PI-PspI | PI-SceI | PluTI |
| PmaCI | PmlI | Ppu21I | PshAI | PsiI | PspCI | PspXI |
| PsrI | PsyI | RgaI | RruI | RsrII | Rsr2I | SacII |
| SapI | SbfI | SdaI | SfaAI | SfiI | SfoI | Sfr274I |
| Sfr303I | SgfI | SgrBI | SgrDI | SgsI | SlaI | SmiI |
| SnaBI | SpeI | SrfI | Sse8387I | SspDI | SstII | SwaI |
| TstI | Tth111I | Vha464I | XagI | XhoI | XmaJI |
 会员登录
会员登录.getTime()%>)
 购物车()
购物车()

 成功收藏产品
成功收藏产品
